遺傳演算法框架 deap 簡介與使用
https://zhuanlan.zhihu.com/p/436438875a
deap框架介紹
目前,有許多可用於遺傳演算法的 Python 框架 —— GAFT,DEAP,Pyevolve 和 PyGMO 等。
其中,deap (Distributed Evolutionary Algorithms in Python) 框架支援使用遺傳演算法以及其他進化計算技術快速開發解決方案,得到了廣泛的應用。deap 提供了各種資料結構和工具,這些資料和工具在實現各種基於遺傳演算法的解決方案時必不可少。
creator模組
creator 模組可以作為元工廠,能夠通過新增新屬性來擴展現有類。
例如,已有一個名為 Employee 的類。使用 creator 工具,可以通過建立 Developer 類來擴展 Employee 類:
import deap import creator
creator.create("Developer",Employee,position="Developer",programmmingLanguage=set)
傳遞給 create() 函數的第一個參數是新類的名稱。第二個參數是要擴展的現有類,接下來是使用其他參數定義新類的屬性。如果為參數分配了一個類(例如 dict 或 set ),它將作為建構函式中初始化的實例屬性新增到新類中。如果參數不是類(例如字串),則將其新增為靜態 (static) 屬性。
因此,建立的 Developer 類將擴展 Employee 類,並將具有一個靜態屬性 position,設定為 Developer,以及一個實例屬性,類型為 set 的 programmingLanguages,該屬性在建構函式中初始化。因此實際上等效於:
class Developer(Employee):
position = "Developer"
def __init__(self):
self.programmmingLanguage = set()
這個新類存在於 creator 模組中,因此需要引用時使用 creator.Developer。
使用 deap 時,creator 模組通常用於建立 Fitness 類以及 Individual 類。
建立適應度類
使用 deap 時,適應度封裝在 Fitness 類中。在 deap 框架中適應度可以有多個組成部分,每個組成部分都有自己的權重(weights)。這些權重的組合定義了適合給定問題的行為或策略。
定義適應度策略
為了快速定義適應度策略,deap 使用了抽象 base.Fitness 類,其中包含 weights 元組,以定義策略並使類可用。可以通過使用 creator 建立基礎 Fitness 類的擴展來完成,類似於建立 Developer 類:
creator.create("FitnessMax",base.Fitness,weights=(1.0,))
上述程式碼將產生一個 creator.FitnessMax 類,該類擴展了 base.Fitness 類,並將 weights 類屬性初始化為 (1.0,)值。需要注意的是:weights 參數是一個元組。
FitnessMax 類的策略是在遺傳演算法過程中最大化單目標解的適應度值。相反,如果有一個單目標問題,需要使適應度值最小的解,則可以使用以下定義來建立最小化策略:
creator.create("FitnessMin",base.Fitness,weights=(-1.0,))
還可以定義具有最佳化多個目標且重要性不同的策略:
creator.create("FitnessCompound",base.Fitness,weights=(1.0,0.2,-0.5))
這將產生一個 creator.FitnessCompound 類,它擁有三個不同的適應度組成部分。第一部分權重為 1.0,第二部分權重為 0.2,第三部分權重為 -0.5。這將傾向於使第一和第二部分(或目標)最大化,而使第三部分(或目標)最小化。
適應度儲存方式
雖然權重元組定義了適應度策略,但是一個對應的元組(稱為 values )用於將適應度值儲存在 base.Fitness 類中。這些值是從單獨定義的函數(通常稱為 evaluate() )獲得的。就像 weights 元組一樣,values 元組儲存每個適應度元件(對象)值。
元組 wvalues 包含通過將 values 元組的每個份量與其 weights 元組的對應份量相乘而獲得的加權值。只要得到了實例的適應度值,就會計算加權值並將其插入 wvalues 中。這些值用於個體之間的適應度的比較操作。
建立個體類
在 deap 中,creator 工具的第二個常見用途是定義構成遺傳演算法種群的個體。遺傳演算法中的個體使用可以由遺傳算子操縱的染色體來表示,通過擴展表示染色體的基類來建立 Individual 類。另外,deap 中的每個個體實例都需要包含其適應度函數作為屬性。
為了滿足這兩個要求,利用 creator 來建立 creator.Individual 類:
creator.create("Individual",list,fitness=creator.FitnessMax)
該程式碼片段具有以下兩個效果:
- 建立的 Individual 類擴展了 Python 的 list 類,這意味著使用的染色體是列表類型
- 建立的 Individual 類的每個實例將具有之前建立的 FitnessMax 屬性
Toolbox類
deap 框架提供的第二種高效建立遺傳演算法的機制是 base.Toolbox 類。Toolbox 用作函數(或操作)的容器,能夠通過別名機制和自訂現有函數來建立新的運算子。
假設有一個函數 sumOfTwo():
def sumOfTwo(a,b):
return a + b
使用 toolbox,可以建立一個新的運算,incrementByFive(),該運算子利用 sumOfTwo() 函數建立:
import base
toolbox = base.Toolbox()
toolbox.register("incrementByFive",sumOfTwo,b=5)
傳遞給 register() 函數的第一個參數是新運算子所需的名稱(或別名),第二個參數是被定製的現有函數。建立完成後,每當呼叫新運算子時,其他參數都會自動傳遞給建立的函數,如:
toolbox.incrementByFive(10)
等效於:
sumOfTwo(10, 5)
這是因為 b 的參數已由 incrementByFive 運算子定義為5。
建立遺傳算子
為了快速建構遺傳流程,可以使用 Toolbox 類定製 tools 模組的現有函數。tools 模組包含許多便捷的函數,這些函數包括選擇、交叉和變異的遺傳算子以及程序的初始化等。
例如,以下程式碼定義了三個別名函數,用作遺傳算子:
from deap import tools
toolbox.register("select", tools.selTournament, tournsize=3)
toolbox.register("mate", tools.cxTwoPoint)
toolbox.register("mutate", tools.mutFlipBit, indpb=0.02)
這三個別名函數的詳細說明:
- select 註冊為 tools 函數 selTournament() 的別名,且 tournsize 參數設定為 3。這將建立 toolbox.select 運算子,其為錦標賽規模為 3 的錦標賽選擇算子
- mate 註冊為 tools 函數 cxTwoPoint() 的別名,這將建立執行兩點交叉的toolbox.mate算子
- mutate 註冊為 tools 函數 mutFlipBit() 的別名,並將 indpb 參數設定為 0.02,這將建立一個翻轉每個特徵的機率為 0.02 的位翻轉突變算子
tools 模組提供了各種遺傳算子的實現,以下列示常用遺傳算子的實現函數。
選擇算子主要包括:
selRoulette() #輪盤選擇
selStochasticUniversalSampling() #隨機遍歷採樣(SUS)
selTournament() #錦標賽選擇
交叉算子主要包括:
cxOnePoint() #單點交叉
cxUniform() #均勻交叉
cxOrdered() #有序交叉
cxPartialyMatched() #實現部分匹配交叉
突變算子主要包括:
mutFlipBit() #位翻轉突變
mutGaussian() #常態分配突變
建立物種
tools 模組的 init.py 檔案包含用於建立和初始化遺傳演算法的函數,其中包括initRepeat(),它接受三個參數:
- 要放置結果對象的容器類型
- 用於生成將放入容器的對象的函數
- 要生成的對象數
如:
#產生含有30個隨機數的列表,這些隨機數介於0和1之間
randomList = tools.initRepeat(list,random.random,30)
此示例中,list 是用作要填充的容器的類型,random.random 是生成器函數,而 30 是呼叫生成器函數以生成填充容器的值的次數。
如果想用 0 或 1 的整數隨機數填充列表,則可以建立一個使用 random.radint() 生成隨機值 0 或 1 的函數,然後將其用作 initRepeat() 的生成器函數:
def zeroOrOne():
return random.randint(0,1)
randomList = tools.initRepeat(list,zeroOrOne,30)
或者,可以利用 Toolbox:
#建立zeroOrOne運算子,使用參數0、1呼叫random.radint()
toolbox.register("zeroOrOne",random.randint,0,1)
randomList = tools.initRepeat(list,tools.zeroOrOne,30)
計算適應度
雖然 Fitness 類定義了確定其策略(例如最大化或最小化)的適應度權重,但實際的適應度是從單獨定義的函數中獲得的。該適應度計算函數通常使用別名 evalidate 來註冊到 Toolbox 模組中:
def someFitnessCalculationFunction(individual):
"""算給定個體的適應度"""
return _some_calculation_of_of_the_fitness(individual)
#將evaluate註冊為someFitnessCalculationFunction()的別名
toolbox.register("evaluate",someFitnessCalculationFunction)
遺傳演算法實踐詳解(deap框架初體驗)
OneMax問題介紹
OneMax 問題是一個簡單的最佳化任務,通常作為遺傳演算法的 Hello World。
OneMax 任務是尋找給定長度的二進制串,最大化該二進制串中數字的總和。例如,對於長度為 5 的OneMax問題,10010 的數字總和為 2,01110 的數字總和為 3。
顯然,此問題的最優解為每位數字均為1的二進制串。但是對於遺傳演算法而言,其並不具備此知識,因此需要使用其遺傳算子尋找最優解。演算法的目標是在合理的時間內找到最優解,或者至少找到一個近似最優解。
遺傳演算法實踐
在進行實踐前,應首先明確遺傳演算法中所用到的要素定義。
- 選擇染色體 由於 OneMax 問題涉及二進制串,因此每個個體的染色體直接利用代表候選解的二進制串表示是一個自然的選擇。在 Python中,將其實現為僅包含 0/1 整數值的列表。染色體的長度與 OneMax 問題的規模匹配。例如,對於規模為 5 的 OneMax 問題,個體 10010 由列表 [1,0,0,1,0] 表示;
- 適應度的計算 由於要最大化該二進制串中數字的總和,同時由於每個個體都由 0/1 整數值列表表示,因此適合度可以設計為列表中元素的總和,例如:sum([1,0,0,1,0])= 2;
- 選擇遺傳算子 選擇遺傳算子並沒有統一的標準,通常可以嘗試幾種選擇方案,找到最適合的方案。其中
選擇算子通常可以處理任何染色體類型,但是交叉和突變算子通常需要匹配使用的染色體類型,否則可能會產生無效的染色體:- 選擇算子:此處選用錦標賽選擇
- 交叉算子:此處選用單點交叉
- 突變算子:此處選用位翻轉突變
- 設定停止條件 限制繁衍的代際數通常是一個不錯的停止條件,它可以確保演算法不會永遠運行。另外,由於我們知道了 OneMax 問題的最佳解決方案(一個全為 1 的二進制串,也就是說其適應度等於代表個體的列表長度),因此可以將其用作另一個停止條件。但是,需要注意的是,對於現實世界中的多數問題而言,通常不存在這種可以公式化精確定義的先驗知識。
遺傳演算法要素組態
在開始實際的遺傳演算法流程之前,需要根據上述要素的設計利用程式碼實現:
- 首先匯入所用包:
from deap import base
from deap import creator
from deap import tools
import random
import matplotlib.pyplot as plt
- 接下來,聲明一些常數,這些常數用於設定 OneMax 問題的參數並控制遺傳演算法的行為:
ONE_MAX_LENGTH = 100 #length of bit string to be optimized
POPULATION_SIZE = 200 #number of individuals in population
P_CROSSOVER = 0.9 #probability for crossover
P_MUTATION = 0.1 #probability for mutating an individual
MAX_GENERATION = 50 #max number of generations for stopping condition
- 接下來,使用 Toolbox 類建立 zeroOrOne 操作,該操作用於自訂 random.randomint(a,b) 函數。通過將參數 a 和 b 固定為值 0 和 1,當在呼叫此運算時,zeroOrOne 運算子將隨機返回 0 或 1:
toolbox = base.Toolbox()#定義toolbox變數
toolbox.register("zeroOrOne",random.randint,0,1)#註冊zeroOrOne運算
- 接下來,需要建立 Fitness 類。由於這裡只有一個目標——最大化數字總和,因此選擇 FitnessMax 策略,使用具有單個正權重的權重元組:
creator.create("FitnessMax",base.Fitness,weights=(1.0,))
- 在 deap 中,通常使用 Individual 的類來代表種群中的每個個體。使用 creator 工具建立該類,使用列表作為基類,用於表示個體的染色體。並為該類增加 Fitness 屬性,該屬性初始化為步驟 4 中定義的 FitnessMax 類:
creator.create("Individual", list, fitness=creator.FitnessMax)
- 接下來,註冊 individualCreator 操作,該操作建立 Individual 類的實例,並利用步驟 1 中自訂的 zeroOrOne 操作隨機填充 0/1。註冊 individualCreator 操作使用基類 initRepeat 操作,並使用以下參數對基類進行實例化:
- 將 creator.Individual 類作為放置結果對象的容器類型
- zeroOrOne 操作是生成對象的函數
- 常數 ONE_MAX_LENGTH 作為要生成的對象數目
由於 zeroOrOne 運算子生成的對像是 0/1 的隨機數,因此,所得的 individualCreator 運算子將使用 100 個隨機生成的 0 或 1 填充單個實例:
toolbox.register("individualCreator",tools.initRepeat,creator.Individual,toolbox.zeroOrOne,ONE_MAX_LENGTH)
- 最後,註冊用於建立個體列表的 populationCreator 操作。該定義使用帶有以下參數的 initRepeat 基類操作:
- 將列表類作為容器類型
- 用於生成列表中對象的函數 —— personalCreator 運算子
這裡沒有傳入 initRepeat 的最後一個參數——要生成的對象數量。這意味著,當使用 populationCreator 操作時,需要指定此參數用於確定建立的個體數:
toolbox.register("populationCreator",tools.initRepeat,list,toolbox.individualCreator)
- 為了簡化適應度(在 deap 中稱為 evaluation )的計算,首先定義一個獨立的函數,該函數接受 Individual 類的實例並返回其適應度。
這裡定義 oneMaxFitness 函數,用於計算個體中 1 的數量。
def oneMaxFitness(individual):
return sum(individual),#deap中的適用度表示為元組,因此,當返回單個值時,需要用逗號將其聲明為元組
- 接下來,將 evaluate 運算子定義為 oneMaxfitness() 函數的別名。使用 evaluate 別名來計算適應度是 deap 的一種約定:
toolbox.register("evaluate",oneMaxFitness)
- 遺傳算子通常是通過對 tools 模組中的現有函數進行別名命名,並根據需要設定參數值來建立的。根據上節設計的要素建立遺傳算子:
toolbox.register("select",tools.selTournament,tournsize=3)
toolbox.register("mate",tools.cxOnePoint)
# mutFlipBit函數遍歷個體的所有特徵,並且對於每個特徵值,
# 都將使用indpb參數值作為翻轉(應用not運算子)該特徵值的機率。
# 該值與突變機率無關,後者由P_MUTATION常數設定。
# 突變機率用於確定是否為種群中的給定個體呼叫mutFlipBit函數
toolbox.register("mutate",tools.mutFlipBit,indpb=1.0/ONE_MAX_LENGTH)
遺傳演算法解的進化
遺傳流程如以下步驟所示:
- 通過使用之前定義的 populationCreator 操作建立初始種群,並以 POPULATION_SIZE 常數作為該操作的參數。並初始化 generationCounter 變數,用於判斷代際數:
population = toolbox.populationCreator(n=POPULATION_SIZE)
generationCounter = 0
- 為了計算初始種群中每個個體的適應度,使用 map() 函數將 evaluate 操作應用於種群中的每個個體。由於 evaluate 操作是 oneMaxFitness() 函數的別名,因此,迭代的結果由每個個體的計算出的適應度元組組成。 得到結果後將其轉換為元組列表:
fitnessValues = list(map(toolbox.evaluate,population)
- 由於 fitnessValues 的項分別與 population (個體列表)中的項匹配,因此可以使用 zip() 函數將它們組合併為每個個體分配相應的適應度元組:
for individual,fitnessValue in zip(population,fitnessValues):
individual.fitness.values = fitnessValue
- 接下來,由於適應度元組僅有一個值,因此從每個個體的適應度中提取第一個值以獲取統計資料:
fitnessValues = [indivalual.fitness.values[0] for individual in population]
- 統計種群每一代的最大適應度和平均適應度。建立兩個列表用於儲存統計值:
maxFitnessValues = []
meanFitnessValues = []
- 遺傳流程的主要準備工作已經完成,在循環時還需設定停止條件,通過限制代際數來設定一個停止條件,而通過檢測是否達到了最佳解(所有二進制串位都為 1 )作為另一個停止條件:
while max(fitnessValues)<ONE_MAX_LENGTH and generationCounter<MAX_GENERATIONS:
- 接下來更新代際計數器:
generationCounter = generationCounter + 1
- 遺傳演算法的核心是遺傳運算子。第一個是 selection 運算子,使用先前利用 toolbox.select 定義的錦標賽選擇。由於我們已經在定義運算子時設定了錦標賽大小,因此只需要將物種及其長度作為參數傳遞給選擇運算子:
offspring = toolbox.select(population,len(population))
- 被選擇的個體被賦值給 offspring 變數,接下來將其克隆,以便我們可以應用遺傳算子而不影響原始種群:
這裡需要注意的是:儘管被選擇的個體被命名為 offspring,但它們仍然是前一代的個體的克隆,我們仍然需要使用 crossover 運算子將它們配對以建立實際的後代。
offspring = list(map(toolbox.clone,offspring)
- 下一個遺傳算子是交叉。已經在上節中定義為 toolbox.mate 運算子,並且其僅僅是單點交叉的別名。使用 Python 切片將 offspring 列表中的每個偶數索引項與奇數索引項對作為雙親。然後,以 P_CROSSOVER 常數設定的交叉機率進行交叉。這將決定這對個體是會交叉或保持不變。最後,刪除後代的適應度值,因為它們現有的適應度已經不再有效:
for child1,child2 in zip(offspring[::2],offspring[1::2]):
if random.random() < P_CROSSOVER:
toolbox.mate(child1,child2)
del child1.fitness.values
del child2.fitness.values
- 最後一個遺傳運算子是突變,先前已註冊為 toolbox.mutate 運算子,並設定為翻轉位突變操作。遍歷所有後代項,將以由突變機率常數 P_MUTATION 設定的機率應用變異算子。如果個體發生突變,我們確保刪除其適應性值。由於該值可能繼承自上一代,並且在突變後不再正確,需要重新計算:
for mutant in offspring:
if random.random() < P_MUTATION:
toolbox.mutate(mutant)
del mutant.fitness.values
- 沒有交叉或變異的個體保持不變,因此,它們的現有適應度值(已在上一代中計算出)就無需再次計算。其餘個體的適應度值為空。使用 Fitness 類的 valid 屬性尋找這些新個體,然後以與原始適應性值計算相同的方式為其計算新適應性:
freshIndividuals = [ind for ind in offspring if not ind.fitness.valid]
freshFitnessValues = list(map(toolbox.evaluate,freshIndividuals))
for individual,fitnessValue in zip(freshIndividuals,freshFitnessValues):
individual.fitness.values = fitnessValue
- 遺傳算子全部完成後,就可以使用新的種群取代舊的種群了:
population[:] = offspring
- 在繼續下一輪循環之前,將使用與上述相同的方法統計當前的適應度值以更新統計資訊:
fitnessValues = [ind.fitness.values[0] for ind in population]
- 獲得最大和平均適應度值,將它們的值新增到統計列表中:
maxFitness = max(fitnessValues)
meanFitness = sum(fitnessValues) / len(population)
maxFitnessValues.append(maxFItness)
meanFItnessValues.append(meanFItness)
print("- Generation {}: Max Fitness = {}, Avg Fitness = {}".format(generationCounter,
maxFitness, meanFitness)
- 此外,使用得到的最大適應度值來找到最佳個體的索引,並列印出該個體:
best_index = fitnessValues.index(max(fitnessValues))
print("Best Individual = ", *population[best_index], "\n")
- 滿足停止條件並且遺傳演算法流程結束後,可以使用獲取的統計資訊,使用matplotlib庫可視化演算法執行過程中的統計資訊,展示各代個體的最佳和平均適應度值的變化:
plt.plot(maxFitnessValues,color='red')
plt.plot(meanFitnessValues,color='green')
plt.xlabel('Generation')
plt.ylabel('Max / Average Fitness')
plt.title('Max and Average fitness over Generation')
plt.show()
該部分完整程式碼如下:
def main():
population = toolbox.populationCreator(n=POPULATION_SIZE)
generationCounter = 0
fitnessValues = list(map(toolbox.evaluate,population))
for individual,fitnessValue in zip(population,fitnessValues):
individual.fitness.values = fitnessValue
fitnessValues = [individual.fitness.values[0] for individual in population]
maxFitnessValues = []
meanFitnessValues = []
while max(fitnessValues) < ONE_MAX_LENGTH and generationCounter < MAX_GENERATION:
generationCounter = generationCounter + 1
offspring = toolbox.select(population,len(population))
offspring = list(map(toolbox.clone,offspring))
for child1,child2 in zip(offspring[::2],offspring[1::2]):
if random.random() < P_CROSSOVER:
toolbox.mate(child1,child2)
del child1.fitness.values
del child2.fitness.values
for mutant in offspring:
if random.random() < P_MUTATION:
toolbox.mutate(mutant)
del mutant.fitness.values
freshIndividuals = [ind for ind in offspring if not ind.fitness.valid]
freshFitnessValues = list(map(toolbox.evaluate,freshIndividuals))
for individual,fitnessValue in zip(freshIndividuals,freshFitnessValues):
individual.fitness.values = fitnessValue
population[:] = offspring
fitnessValues = [ind.fitness.values[0] for ind in population]
maxFitnessValue = max(fitnessValues)
meanFitnessValue = sum(fitnessValues) / len(population)
maxFitnessValues.append(maxFitnessValue)
meanFitnessValues.append(meanFitnessValue)
print("- Generation {}: Max Fitness = {}, Avg Fitness = {}".format(generationCounter,maxFitnessValue,meanFitnessValue))
best_index = fitnessValues.index(max(fitnessValues))
print("Best Indivadual = ", *population[best_index],"\n")
plt.plot(maxFitnessValues,color="red")
plt.plot(meanFitnessValues,color="green")
plt.xlabel("Generation")
plt.ylabel("Max / Average Fitness")
plt.title("Max and Average fitness over Generation")
plt.show()
至此可以開始測試我們的遺傳演算法了,運行程式碼以驗證其是否找到了 OneMax 問題的最優解。
演算法運行
if __name__ == "__main__":
main()
運行程序時,可以看到程式執行輸出:
- Generation 27: Max Fitness = 99.0, Avg Fitness = 96.805
Best Indivadual = 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
- Generation 28: Max Fitness = 99.0, Avg Fitness = 97.235
Best Indivadual = 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
- Generation 29: Max Fitness = 99.0, Avg Fitness = 97.625
Best Indivadual = 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
- Generation 30: Max Fitness = 100.0, Avg Fitness = 98.1
Best Indivadual = 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
可以看到 30 代後演算法找到全 1 解,結果適應度為 100,並停止了遺傳流程。平均適應度開始時僅為 53 左右,結束時接近 100。
繪製圖形如下所示:
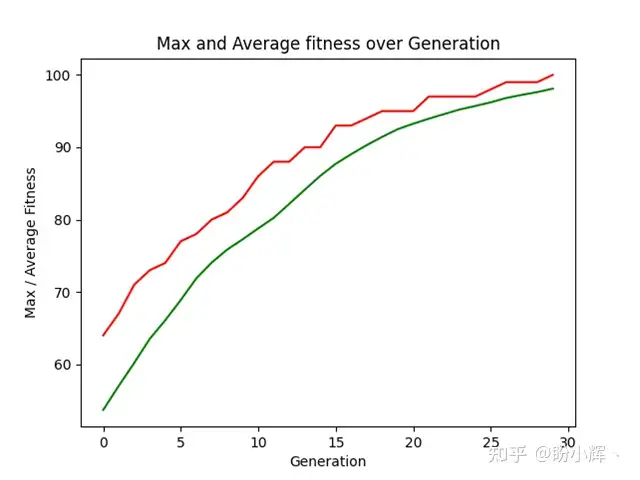
適應度變化
該圖說明瞭最大適應度與平均適應度是如何隨著代數的增加而逐步增加。